| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
| 29 | 30 | 31 |
- 데이터분석
- DATA
- 리눅스
- 텍스트마이닝
- 텍스트분석
- pytorch
- YarinGal
- Graph
- 파이썬
- 코딩테스트
- 불확실성
- 논문리뷰
- 강화학습
- dropout
- 우분투
- R
- 백준
- 크롤링
- 베이지안
- Crawling
- GNN
- AI
- 알고리즘
- selenium
- bayesian
- uncertainty
- PYTHON
- VAE
- pandas
- 빅데이터
- Today
- Total
끄적거림
data.table 패키지 유용하게 사용하기 1편 본문
나는 원래 R 사용자였으며, 나름 학부시절 잘하는 쪽에 속한다고 생각했다.
그것이 가능했던 것이 data.table 패키지를 어느정도 잘 사용하고 나서부터라고 생각한다.
그동안 내가 알고 있던 꿀팁들을 적어볼까한다.
1. data.table 패키지 소개
여기저기 찾아보면 많은 자료들이 있으니 간단하게 소개하고 넘어가겠다.
우선 기능적으로 보았을 때,
1) 빠른 계산력을 제공한다.
가장 기본적인 매트릭스 형태로 R에서는 data.frame을 제공한다. 하지만, 사용하다보면 가독성도 떨어지고, 사용하기 불편하고, 다소 느린 단점들이 있다.
이런 부분들을 개선한 것이 data.table이다. 데이터 성격에 따라 차이가 있겠지만 대략 10~100배 정도 빠르다고 생각하면 된다.
2) 적용 범위가 넓다.
data.table은 data.frame 객체를 포함하고 있게 때문에 data.frame을 대상으로한 함수나 설정들도 똑같이 작용한다.
3) 강력한 함수 fread, fwrite
python과 R, 기타 다른 통계 패키지에서 데이터를 읽고 불러드리는데 가장 빠른 것이 바로 data.table의 fread함수이다. 이에 대한 자세한 얘기는 나중에 하기로 하자.
2. 조건 걸기, 연산하기, 그룹핑을 한 번에!
data.table이 된 객체는 인덱싱처리와 비슷해보이는 기능들을 제공한다.
install.packages("data.table")
library(data.table)
data <- as.data.table(data)위와 같이 data라는 data.table 객체가 새로 생성되었다고 하자.
이때, data라는 객체는 대괄호 : [ ] 를 이용하여 다음과 같은 기능을 실행시킬 수 있다.

1) I영역 : 조건 걸기
I영역을 이용하여 전체 데이터에서 filtering을 걸 수 있다.
iris <- as.data.table(iris)
iris[Sepal.Length > 4]가장 친숙한 iris 데이터로 예를 들면, 위와 같은 코드는 Sepal.Length라는 변수 중 4보다 큰 row들을 뽑아 낼 수 있다.
이것은 tidyverse 패키지의 filter() 함수와 같은 기능을 갖는다.
다만, tidyverse 패키지의 filter() 함수와 다른 점은 데이터 셋 자체에서 필터링 할 수 있다는 점이다.
이해가 안 갈 수 있으니 엑셀을 예로 생각해보자.
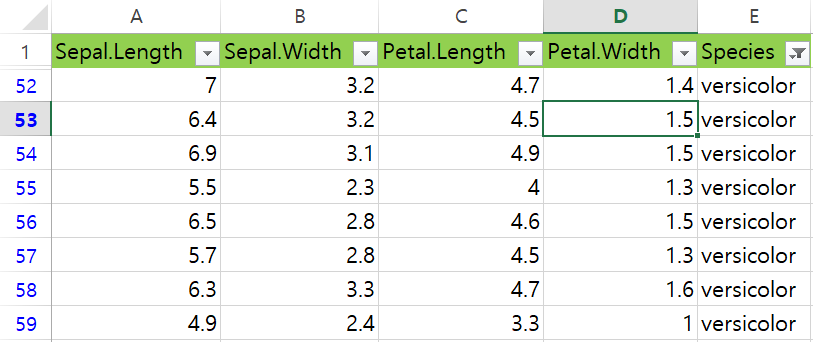
그림과 같이 엑셀에서 Species == "versicolor"인 조건을 걸어 필터링 했을 때의 모습이다.
이것이 data.table에서의 조건 걸기와 같은 느낌이다.
하지만, tidyverse 패키지의 filter()함수나 기타 다른 필터링이 가능한 함수들은 조건을 걸고 나온 데이터들을 다른 객체에 임시로 저장하여 보여준다.
이것의 차이는 어마어마하게 크다. 큰 이유는 J영역에서 살펴보기로 하자.
2) J영역 : 연산하기
J영역에서는 여러 연산을 컬럼별로 할 수 있다.
iris[, sum(Sepal.Length)]이 코드를 입력하면 sum(iris$Sepal.Length)와 같은 결과값이 도출된다. 기본적인 집계함수 뿐만 아니라 컬럼 생성 연산, counting 연산, 기타 대부분의 함수들이 적용된다.(단, parameter가 둘 이상인 경우는 특수하게 적용해야한다.)
위 1)번에서 중요하다고 언급한 부분을 다음 코드를 보며 이해해보자.
iris[Species == "setosa", isLarge := ifelse(Sepal.Length >= 4, "Large", "Normal")]이 코드는 꽃의 종이 setosa인 경우에 한하여, Sepal.Length가 4이상인 경우 Large, 아니면 Normal이라는 값을 isLarge 컬럼에 ":=" 연산자를 통하여 새로 만들었다.
이 코드가 실행되고 나면 Species != "setosa" 인 경우(꽃의 종이 setosa가 아닌 경우)는 어떻게 될까?
정답은 Null값으로 찍힌다. 조건을 setosa만 걸었고, 그에 한하여 연산을 진행한 결과를 새로운 컬럼에 넣었기 때문에 나머지 꽃의 종과는 무관한 것이다. 그렇기 때문에 data.table 패키지는 null값을 넣어 새로운 컬럼을 채운다.
만약 이를 tidyver 패키지로 표현하려면 어떻게 할 수 있을까?
iris <- iris %>%
filter(Species == "setosa") %>%
mutate(isLarge = ifelse(Sepal.Length >= 4, "Large", "Normal")) %>%
setDT()이렇게 하면 동일한 결과를 도출할 수 있을까? 아니다. 위 코드를 실행하면 setosa가 아닌 종들의 데이터들은 증발된다. 제대로 된 결과를 도출하기 위해서는 다음과 같다.
iris <- iris %>%
mutate(isLarge = ifelse(Species == "setosa",
ifelse(Sepal.Length >= 4, "Large", "Normal"), null)) %>%
setDT()
data.table 패키지와 tidyverse 패키지를 이용하여 같은 결과를 가져왔지만, 사뭇 다른 느낌이다.
이 두 코드의 성능 측면이나 가독성 측면에서 data.table 패키지가 더 낫다는 것을 알 수 있다.
3) K영역 : 그룹핑
사실 K영역에서는 그룹핑 말고 더 많은 기능을 제공한다. 하지만 개인적으로 data.table을 사용하다보면 grouping 이외의 기능은 잘 사용하지 않기때문에 grouping에 대해서만 얘기해보자.
iris[, sum(Sepal.Length), by = list(Species)]모두가 느끼겠지만, 각 Species 별로 Sepal.Length의 합계를 도출하는 코드이다. 여기서 꿀팁 중 꿀팁이 list()함수를 사용하면 컬럼에 직접 접근할 수 있다. 여러 컬럼에 의해 grouping을 진행할 계획이면 list()함수를 추천한다. 그렇지 않을 경우, 벡터 형태(c("A", "B")) 이런 식으로 컬럼명을 string 형태로 바꾸어 인자값으로 전달해야한다.
'R쓸신잡' 카테고리의 다른 글
| [R쓸신잡] multi package install, import(library), update in R (0) | 2020.06.19 |
|---|---|
| [R쓸신잡]DB연결 in R 패키지(RJDBC, RODBC, ROracle) benchmark (0) | 2020.06.18 |
| [R쓸신잡]DB연결 in R with ROracle package (0) | 2020.06.17 |
| [R쓸신잡]DB연결 in R with RODBC package (0) | 2020.06.16 |
| [R쓸신잡]DB연결 in R with RJDBC package (1) | 2020.06.15 |
